環境研究総合推進費 (JPMEERF20204004)
令和4年3月31日終了
農学研究院 荒木 仁志 教授( 動物生態学研究室)
背景・目的
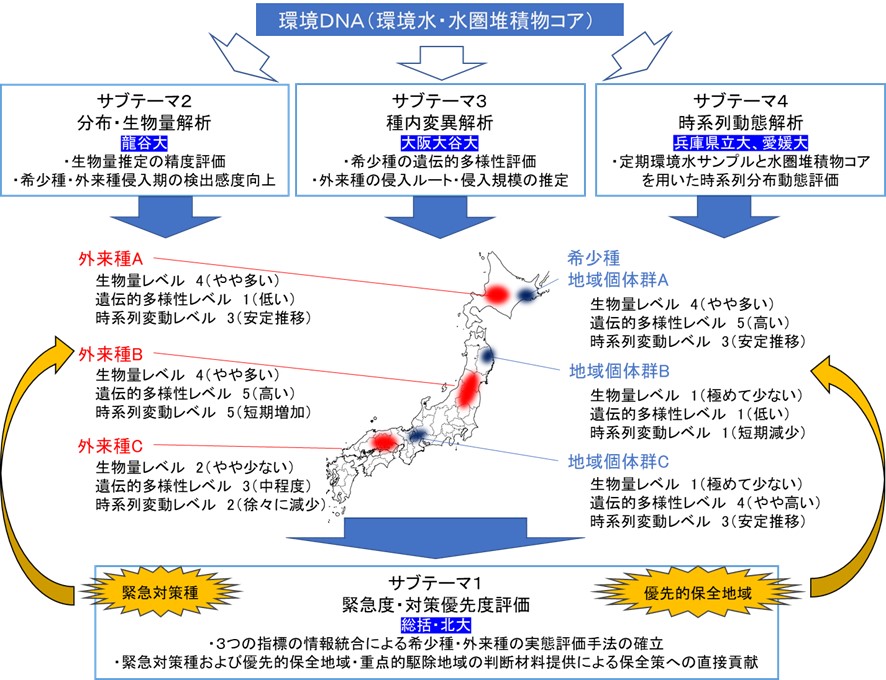
生態系サービスを支える生物多様性と地域生態系の健全な維持管理は、国連が掲げる持続可能な開発目標(SDGs)の根幹を成している。しかしながら、捕獲調査に基づく現行の生物多様性評価手法は経費・労力等の制約から局所的・限定的にならざるを得ず、希少種・外来種対策の適切な優先度選定を困難にしている。
近年生態学ツールとして脚光を浴びる「環境DNA技術」は、この問題への有効な解決手段となり得る。環境DNAとは、環境媒体(河川水、土壌、空気等)に含まれる生物由来のDNAを指す。環境DNAを用いて野生生物の実態を捉えることが出来れば、発見や捕獲が困難な希少種・外来種の置かれた状況をより迅速に把握することが可能となり、希少種の絶滅や外来種拡散の予防に役立つ。本プロジェクトでは、種レベルでの希少種・外来種の生物量推定精度の検証と手法改良に加え、環境DNAを用いて種内変異と集団遺伝構造を解析する技術を開発し、希少種の遺伝的多様性評価や外来種の侵入規模推定を可能にすることを目指す。
更に従来の環境水に加え、より長期的な生物動態を反映し得る水圏堆積物コアを用いた環境DNA解析を組み合わせることにより、時系列に沿った個体群動態情報を入手可能となることが期待される。これらの方法で入手した生物量、遺伝的多様性と時系列動態の情報統合を行うことで、希少種の絶滅リスクや外来種分布拡大等の実態をより効率的かつ正確に把握でき、緊急対策を要する種や保全・駆除優先地域をより的確に選定することが可能となる。
「研究内容(計画・構想)」および「期待される成果」
本研究プロジェクトの最終目標は、環境DNA技術を用いて希少種・外来種の分布・動態や種内変異に関する情報を多次元的に収集し、それらを統合することで個々の希少種・外来種問題に対する対策優先度を科学的に評価する方法を提案することにある。環境DNA技術を用いた希少種の研究手法についてはすでに検討が始まりつつあるが、種内変異や時系列を含めた解析により、希少種保全の優先度評価がより頑強かつ明確になることが期待される。また外来種についても環境水・水圏堆積物コアサンプル由来の環境DNA解析を組み合わせることにより、既存の環境DNA技術では把握できなかった時系列動態に関する情報を得ることが出来る。そのため、希少種・外来種に特化した生物量推定手法の精度検証および改良を実施しつつ、これを種内変異に応用した集団遺伝解析、同所的な環境水・水圏堆積物コアサンプルの分析からの時系列解析を行い、これらを統合した対策優先度評価手法の確立を目指す。加えて北海道や本州における実践研究を行い、これら最新技術の応用と実地検証を試みることで、希少種・外来種問題への具体的な対策に資する研究成果も期待される。
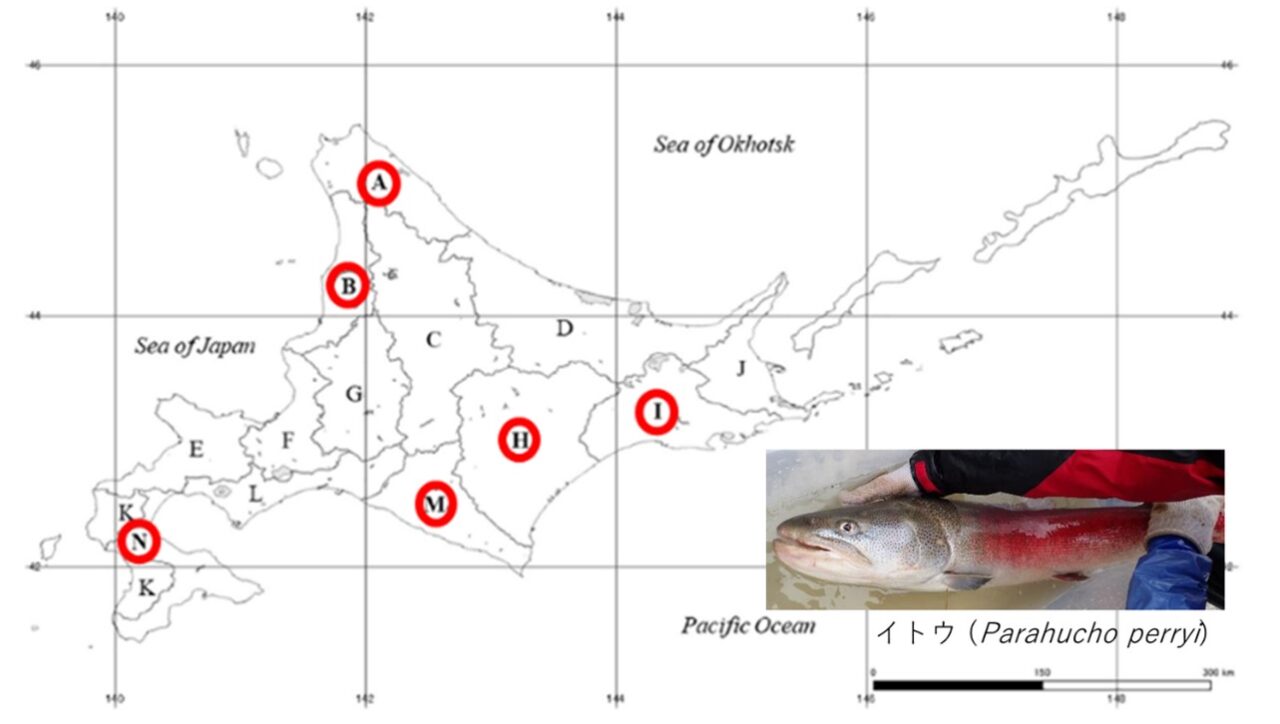
1) 絶滅危惧種イトウの環境DNAが検出された7河川を含む地域。A地域のみ2河川から、その他赤丸の地域では各1河川からイトウDNAが検出された(Mizumoto et al. 2020 Front. Ecol. Evol.より改変)
2) 魚類捕獲調査風景

3) 河川環境測定

4) 夜間の環境DNA採集風景

5) ドローンを用いた環境DNA採集風景

6) 環境DNAろ過風景

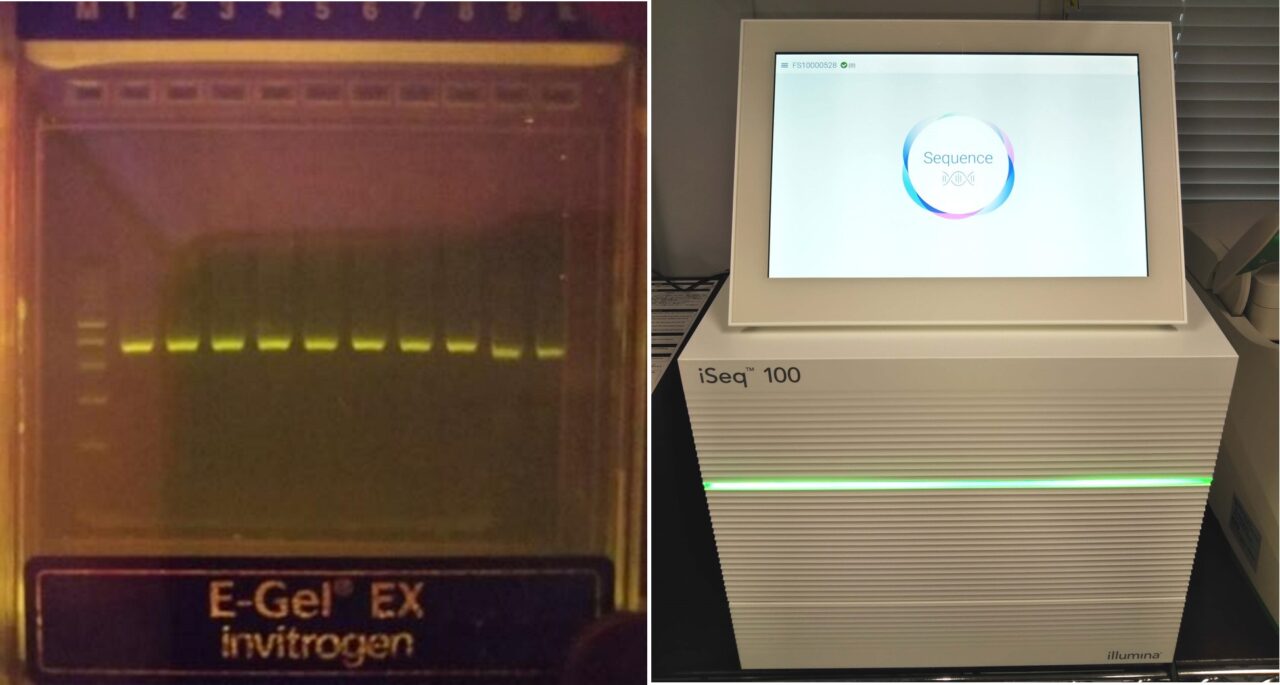
7) E-Gel電気泳動システムにより確認した環境DNAのPCR増幅(左)と環境DNA解析に用いた次世代シーケンサー iSeq 100(右)
研究プロジェクトメンバー
- 研究代表者
荒木 仁志 教授
北海道大学大学院農学研究院・動物生態学研究室 - 研究分担者
山中 裕樹 准教授
龍谷大学先端理工学部環境生態工学課程内井 喜美子 助教
谷 佳津治 教授
見坂 武彦 准教授
大阪大谷大学薬学部衛生・微生物学講座土居 秀幸 准教授
兵庫県立大学大学院情報科学研究科加 三千宣 准教授
愛媛大学沿岸環境科学研究センター
成果概要
環境再生保全機構の環境研究総合推進費中間評価のリンクです。ご参照ください。
自然共生領域、環境問題対応型研究 4-2004
中間評価個票
成果概要
研究成果の発表状況
1)H. MIZUMOTO, O. KISHIDA, K. TAKAI, N. MATSUURA and H. ARAKI. Biological Invasions 24:1199-1211 (2022)
Utilizing environmental DNA for wide-range distributions of reproductive area of an invasive terrestrial toad in Ishikari river basin in Japan.
https://link.springer.com/article/10.1007/s10530-021-02709-y
プレスリリース:https://www.hokudai.ac.jp/news/2021/12/post-958.html
2) H. ARAKI, T. KANBE, H. YAMANAKA, Y. MINEGISHI and N. KOIZUMI. Environmental DNA 4:487-491 (2022)
edna2021: The fourth annual meeting of The eDNA Society “Species distributions, and beyond”
https://onlinelibrary.wiley.com/doi/full/10.1002/edn3.278
3)T. MINAMOTO, M. MIYA, T. SADO, S. SEINO, H. DOI, M. KONDOH, K. NAKAMURA, T. TAKAHARA, S. YAMAMOTO, H. YAMANAKA, H. ARAKI, W. IWASAKI, A. KASAI, R. MASUDA and K. UCHII. Environmental DNA 3: 8-13 (2021)
An illustrated manual for environmental DNA research: Water sampling guidelines and experimental protocols.
https://onlinelibrary.wiley.com/doi/full/10.1002/edn3.121
4)T. YATSUYANAGI and H. ARAKI. Plos ONE 15: e0239912 (2020)
Understanding seasonal migration of Shishamo smelt in coastal regions using environmental DNA.
https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0239912
5)H. MIZUMOTO, T. MITSUZUKA and H. ARAKI. Front. Ecol. Evol. 8: 569425 (2020)
An environmental DNA survey on distribution of an endangered salmonid species, Parahucho perryi, in Hokkaido, Japan.
https://www.frontiersin.org/articles/10.3389/fevo.2020.569425/full
プレスリリース:https://www.hokudai.ac.jp/news/2020/11/post-749.html
6)H. DOI, T. MIMAMOTO, T. TAKAHARA, S. TSUJI, K. UCHII, S. YAMAMOTO, I. KATANO, H. YAMANAKA. Ecological Research 36: 379-388 (2021)
Compilation of real-time PCR conditions toward the standardization of eDNA methods.
https://esj-journals.onlinelibrary.wiley.com/doi/abs/10.1111/1440-1703.12217
7)H. DOI, R. INUI, S. MATSUOKA, Y. AKAMATSU, M. GOTO and T. KONO. Freshwater Biology 66:1257-1266 (2021)
Estimation of biodiversity metrics by environmental DNA metabarcoding compared with visual and capture surveys of river fish communities.
https://onlinelibrary.wiley.com/doi/abs/10.1111/fwb.13714
8)T. SAITO and H. DOI. Front. Ecol. Evol. 9:623831 (2021)
A model and simulation of the influence of temperature and amplicon length on environmental DNA degradation rates: A meta-analysis approach.
https://www.frontiersin.org/articles/10.3389/fevo.2021.623831/full
9)H. DOI, T. WATANABE, N. NISHIZAWA, T. SAITO, H. NAGATA, Y. KAMEDA, N. MAKI, K. IKEDA, and T. FUKUZAWA. Mol. Ecol. Res. 21:2364-2368 (2021)
On-site environmental DNA detection of species using ultra-rapid mobile PCR.
https://onlinelibrary.wiley.com/doi/abs/10.1111/1755-0998.13448
10) 一般社団法人 環境DNA学会 企画・ 土居、近藤 編 共立出版(2021)
「環境DNA -生態系の真の姿を読み解く-」
https://www.kyoritsu-pub.co.jp/book/b10003313.html
11) K. UCHII, K. WAKIMURA, T. KIKKO, R. YONEKURA, R. KAWAGUCHI, H. KOMADA, H. YAMANAKA, T. KENZAKA, K. TANI. Limnology 23: 49–56 (2022) Environmental DNA monitoring method of the commercially important and endangered fish Gnathopogon caerulescens.
https://onlinelibrary.wiley.com/doi/abs/10.1111/1755-0998.13448
12) N. TSUGEKI, K. NAKANE, H. DOI, N. OCHI and M. KUWAE. Scientific Reports 12:1741 (2021)
Reconstruction of 100-year dynamics in Daphnia spawning activity revealed by sedimentary DNA.
https://www.nature.com/articles/s41598-021-03899-0
13) Y. SEGAWA, M. YAMAMOTO, M. KUWAE, K. MORIYA, H. SUZUKI, K. SUZUKI. Journal of Geophysical Research: Biogeosciences 127: e2022JG006825 (2022)
Reconstruction of the Eukaryotic Communities in Beppu Bay over the past 50 years based on ssedimentary DNA barcoding.
https://doi.org/10.1029/2022JG006825
14) T. JO and H. YAMANAKA. Freshw. Biol. Online first (2022)
Meta-analyses of environmental DNA downstream transport and deposition in relation to hydrogeography in riverine environments.
https://onlinelibrary.wiley.com/doi/full/10.1111/fwb.13920